近日,华南师范大学环境学院应光国教授团队石义静副教授在《Environmental Science & Technology》上发表了题为“Soil Amoebae Are Unexpected Hotspots of Environmental Antibiotics and Antibiotic Resistance Genes”的论文(https://url.scnu.edu.cn/record/view/index.html?key=9391b5dc5fc5c952dbcba6222b15a0f0)。

图文摘要
全文速览
研究探讨了原生生物,特别是阿米巴与抗生素的相互作用及其对抗生素抗性基因(ARGs)和金属抗性基因(MRGs)命运的影响。研究发现,阿米巴对八种高风险环境抗生素表现出高抗性,并能在细胞内积累大量抗生素。不同地区的野生阿米巴菌株携带了大量的ARGs和MRGs,且在同一物种内部存在显著的异质性。阿米巴共生菌和病原体主要携带这些基因。与自由生活的菌株相比,阿米巴共生菌Paraburkholderia的基因组较小,抗性基因较少;而阿米巴携带的环境致病菌Stenotrophomonas maltophilia并没有显示出显著减少的基因组大小、且含有大量抗性基因。研究揭示土壤阿米巴是抗生素和抗性基因的意外热点,并建议将原生生物纳入更广泛的抗生素抗性研究中。
引言
抗生素抗性对人类健康构成重大威胁,导致全球每年大量死亡。抗生素抗性基因(ARGs)和共定位的金属抗性基因(MRGs)的传播是抗生素抗性的主要原因,这一过程因抗生素的过度使用而加速。目前,大多数关于ARGs和MRGs的研究集中在细菌上,而对于抗生素与其他重要微生物群、如原生生物的相互作用知之甚少。原生生物是土壤中无处不在且多样性丰富的单细胞真核生物,它们在土壤微生物群落中常被忽视。作为细菌、真菌和线虫的消费者,异养原生生物在调节土壤微生物群落中起着关键作用。原生生物作为有效的微生物捕食者,对细菌施加强烈的选择压力,有证据表明原生生物可以与抗生素相互作用,影响抗生素抗性的传播和出现。许多细菌能在原生生物内生存,特别是变形虫,使原生生物成为潜在的病原体载体。
图文导读
研究首先发现抗生素暴露影响阿米巴的发育,但不影响它们的孢子总量,所有8种高风险环境抗生素对阿米巴的生长几乎没有影响。然而,形态计量学分析表明,抗生素暴露显著影响阿米巴的发育。目前尚不清楚为什么抗生素会引起阿米巴的这种表型变化,但先前的研究表明阿米巴的茎长缩短与应激反应有关。因此,这些结果表明,尽管抗生素暴露不会影响阿米巴的总孢子产量,但它们仍然会引起土壤阿米巴的应激反应。
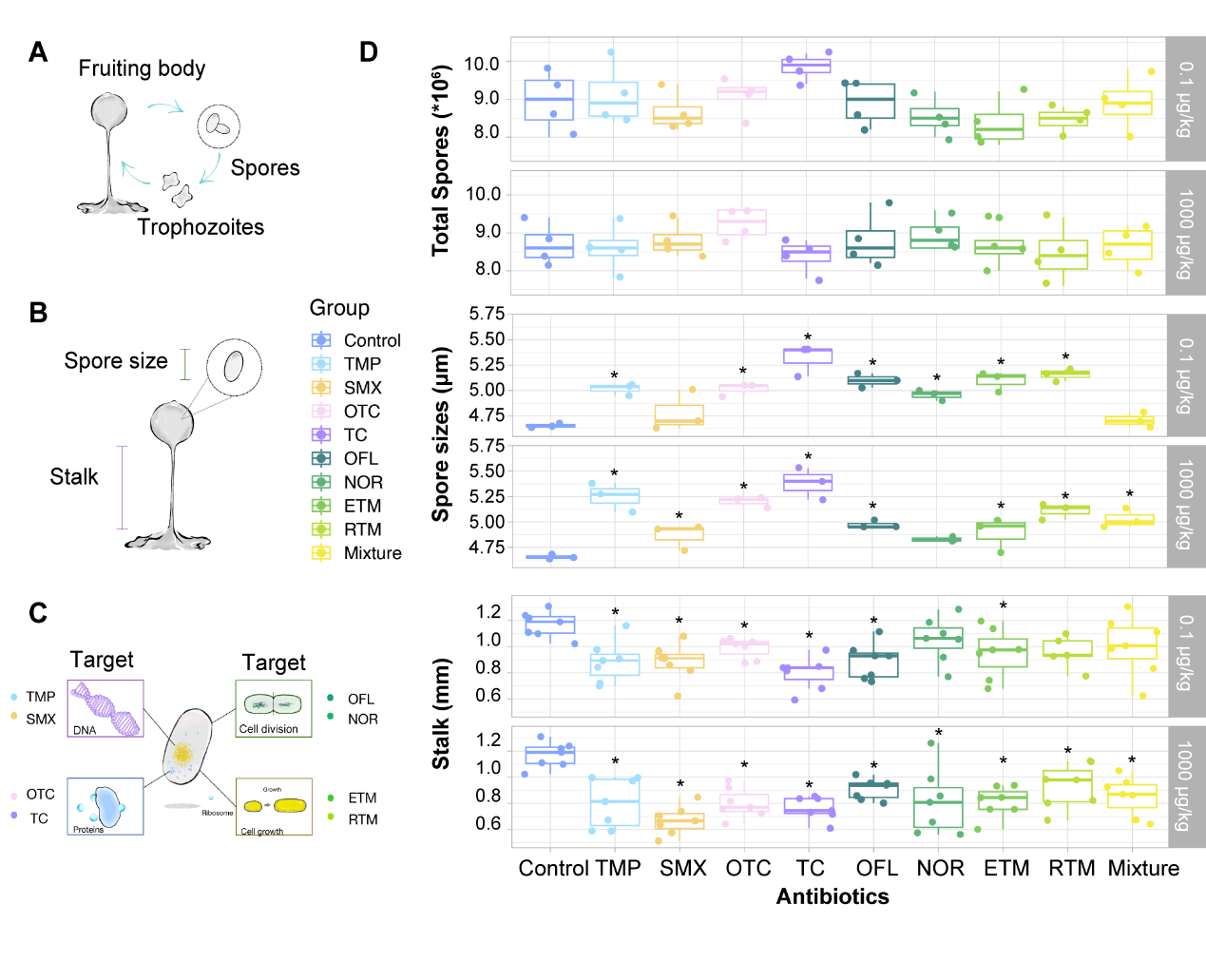
Figure 1. A) Life cycle of the social amoeba D. discoideum. B) Illustration of the fruiting body showing stalk length and spore size. C) Eight high-risk environmental antibiotics and their mechanism of action. D) Total spore production, spore size, and stalk length of amoebae in response to eight antibiotics. Abbreviations: Trimethoprim (TMP), Sulfamethoxazole (SMX), Oxytetracycline (OTC), Tetracycline (TC), Ofloxacin (OFL), Norfloxacin (NOR), Erythromycin (ETM), and Roxithromycin (RTM). The asterisks represent significant differences between antibiotic treatment and control, based on ANOVA and Fisher’s Least Significant Difference (LSD) test (p < 0.05).
鉴于土壤阿米巴对抗生素的高抗性,我们随后的研究重点是抗生素在其体内的生物积累和命运,发现阿米巴在细胞内积累大量抗生素。同时,还发现阿米巴主动消除其生殖孢子中过量的抗生素,从而防止过量的抗生素跨代进入其后代细胞。
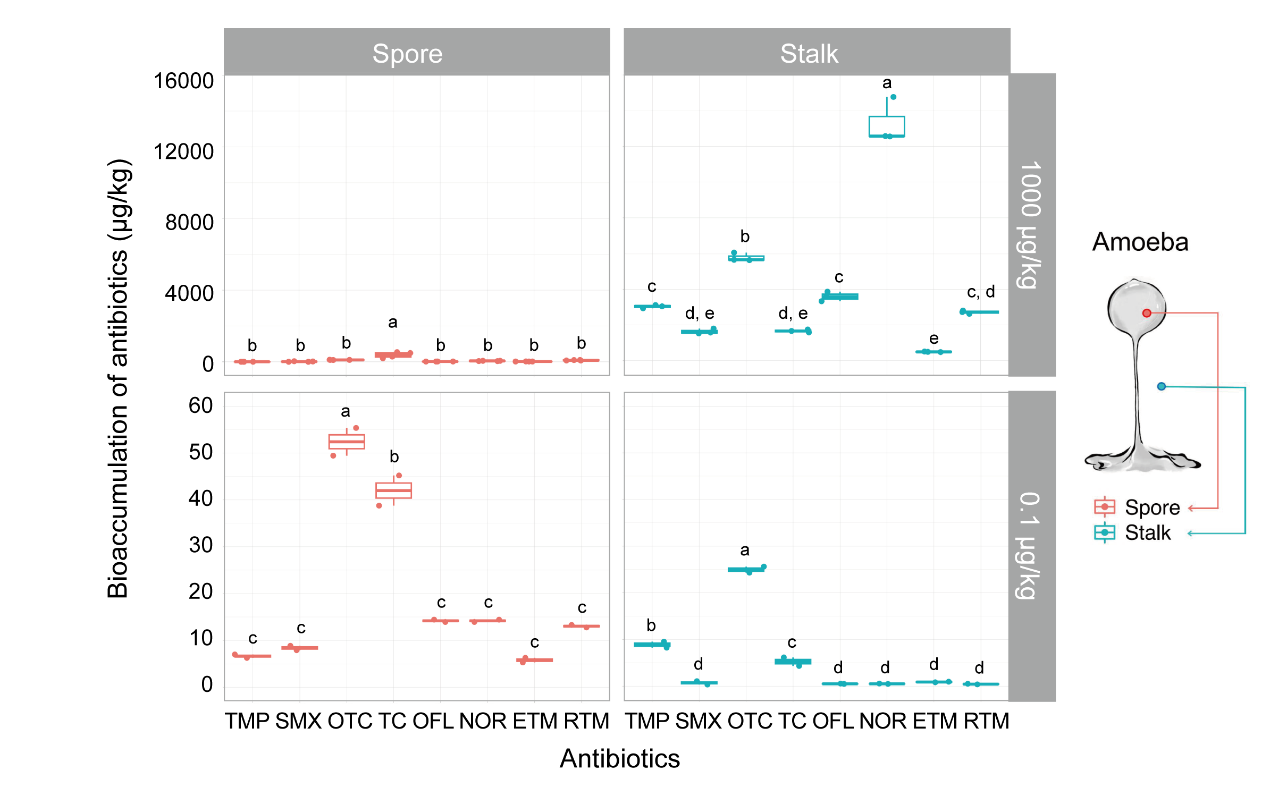
Figure 2. Bioaccumulation and transgenerational passage of antibiotics in amoebae. Abbreviations: Trimethoprim (TMP), Sulfamethoxazole (SMX), Oxytetracycline (OTC), Tetracycline (TC), Ofloxacin (OFL), Norfloxacin (NOR), Erythromycin (ETM), and Roxithromycin (RTM). Different lowercase letters represent significant differences between factor levels, based on GLM analysis and Fisher’s Least Significant Difference (LSD) test (p < 0.05).
发现土壤阿米巴是ARGs和MRGs的环境热点,野生阿米巴菌株携带大量的ARGs和MRGs,且在单一物种内部存在显著的异质性。在分离的阿米巴株中共鉴定出142个ARGs和170个MRGs,各分为13种类型。
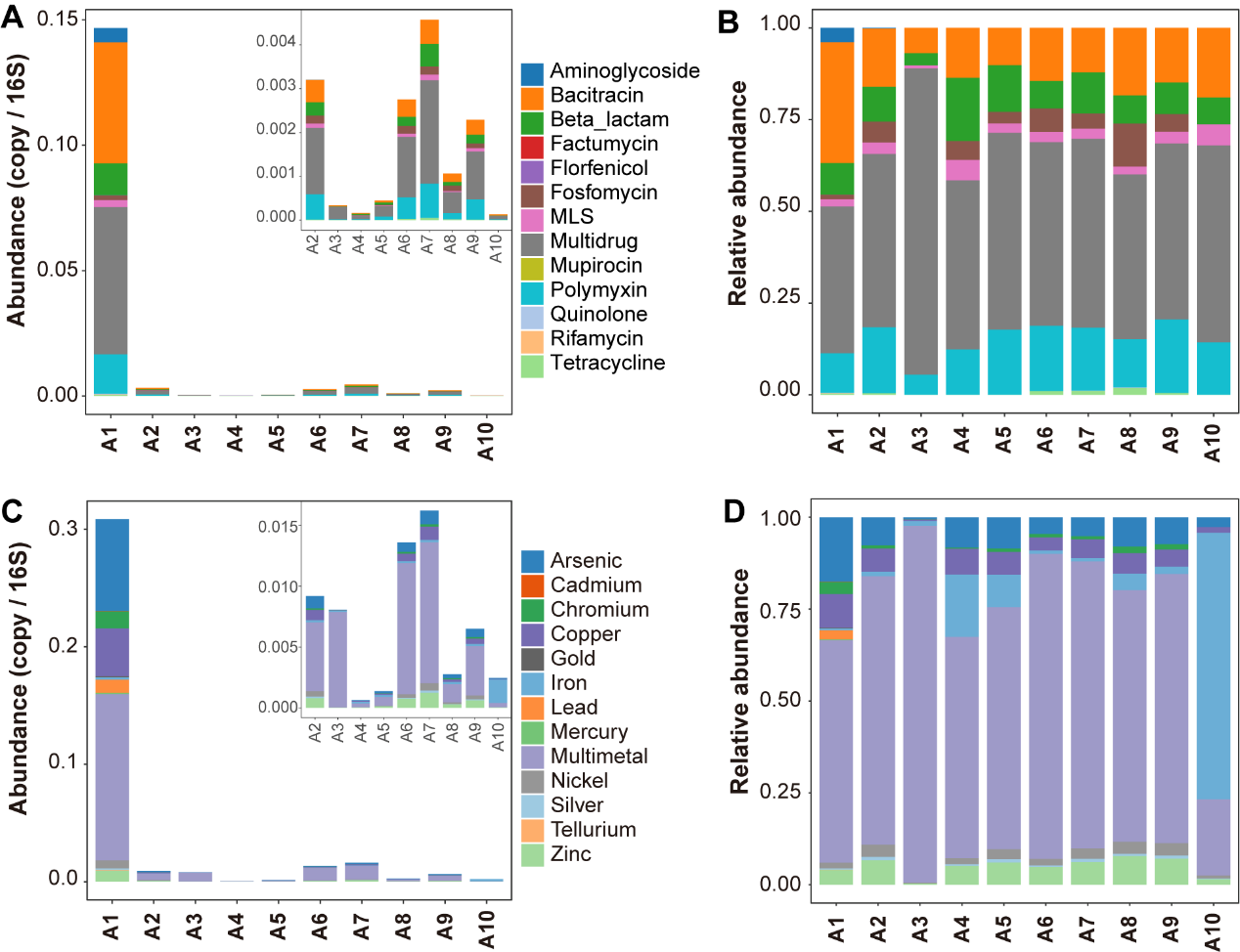
Figure 3. The abundance (A, C) and relative abundance (B, D) of antibiotic and metal resistance genes in wild amoeboid strains.
阿米巴相关细菌携带了大多数抗性基因。总的来说,ARGs的主要宿主包括副霍尔德氏菌、黑葡萄球菌、新葡萄球菌、真菌假单胞菌、嗜麦芽窄养单胞菌和大肠杆菌。值得注意的是,一些临床病原体也被确定为宿主,并在所有分离的阿米巴菌株中普遍存在。
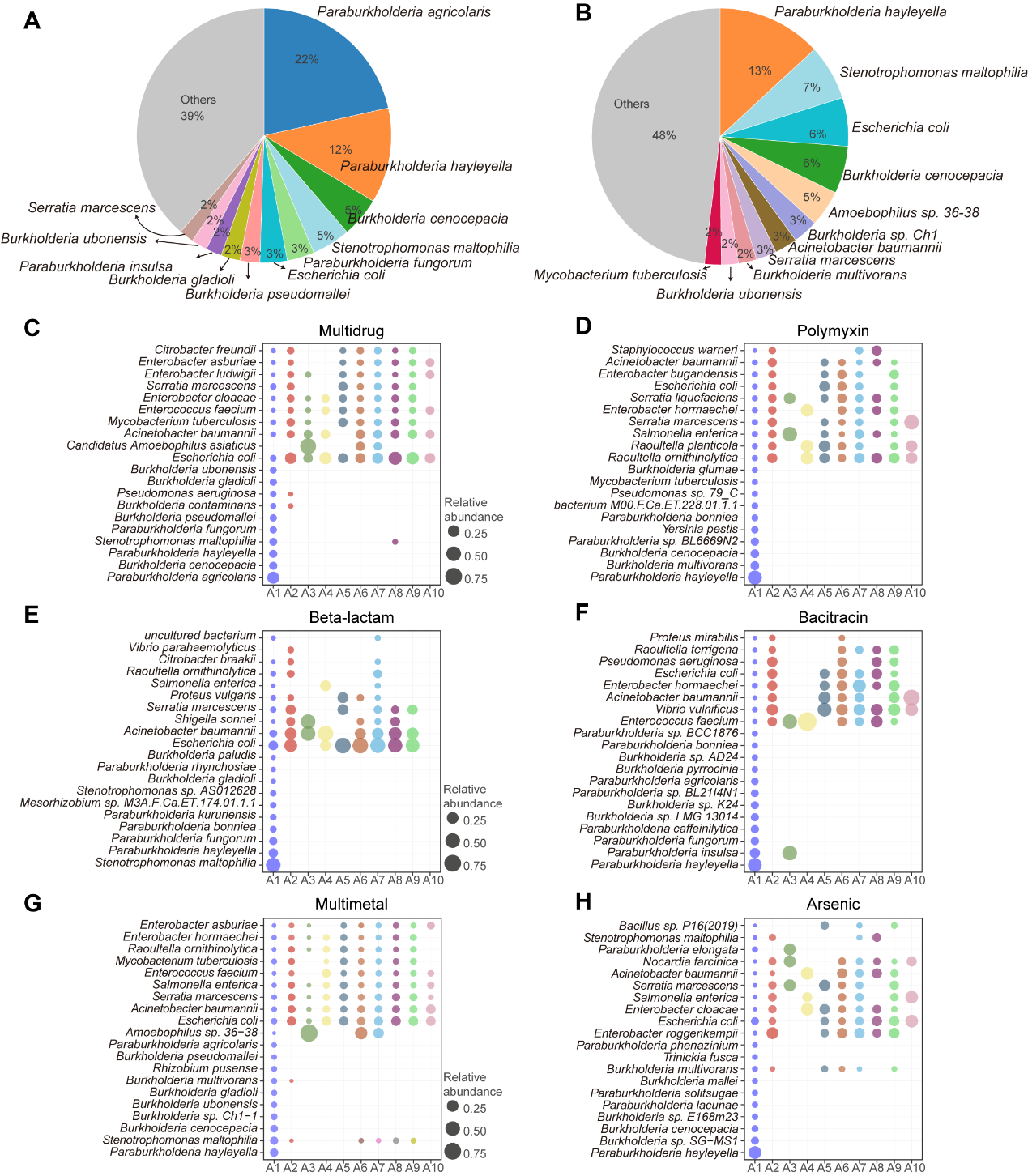
Figure 4. Bacterial hosts of the antibiotics (A) and metal resistance (B) genes in amoebae. Distribution of resistance genes in different bacterial and amoeba hosts: (C) multidrug; (D) polymyxin; (E) beta-lactam; (F) bacitracin; (G) multimetal; (H) arsenic.
与自由生活的菌株相比,阿米巴共生细菌(如Paraburkholderia symbionts)的基因组减少,含有的抗性基因也较少。例如,P. hayleyella和P. bonniea共丢失了6个ARGs (amrAB、Omp38、ceoB、opcM和qacE), 6个MRGs (tr、cnrT、czcR、hmrR、mdtB/yegN和mntP/yebN), 2个移动遗传元件(tniB和tnpA1)和11个毒力因子(VFs) (pilA、PA2359、tssB-5、tssC、galE、gmd、kdsA、rfbE、flhC、cdpA和katG)。这表明,随着共生时间的增加,阿米巴细胞内共生菌表现出基因组缩小的现象,同时伴随着抗性基因的减少。
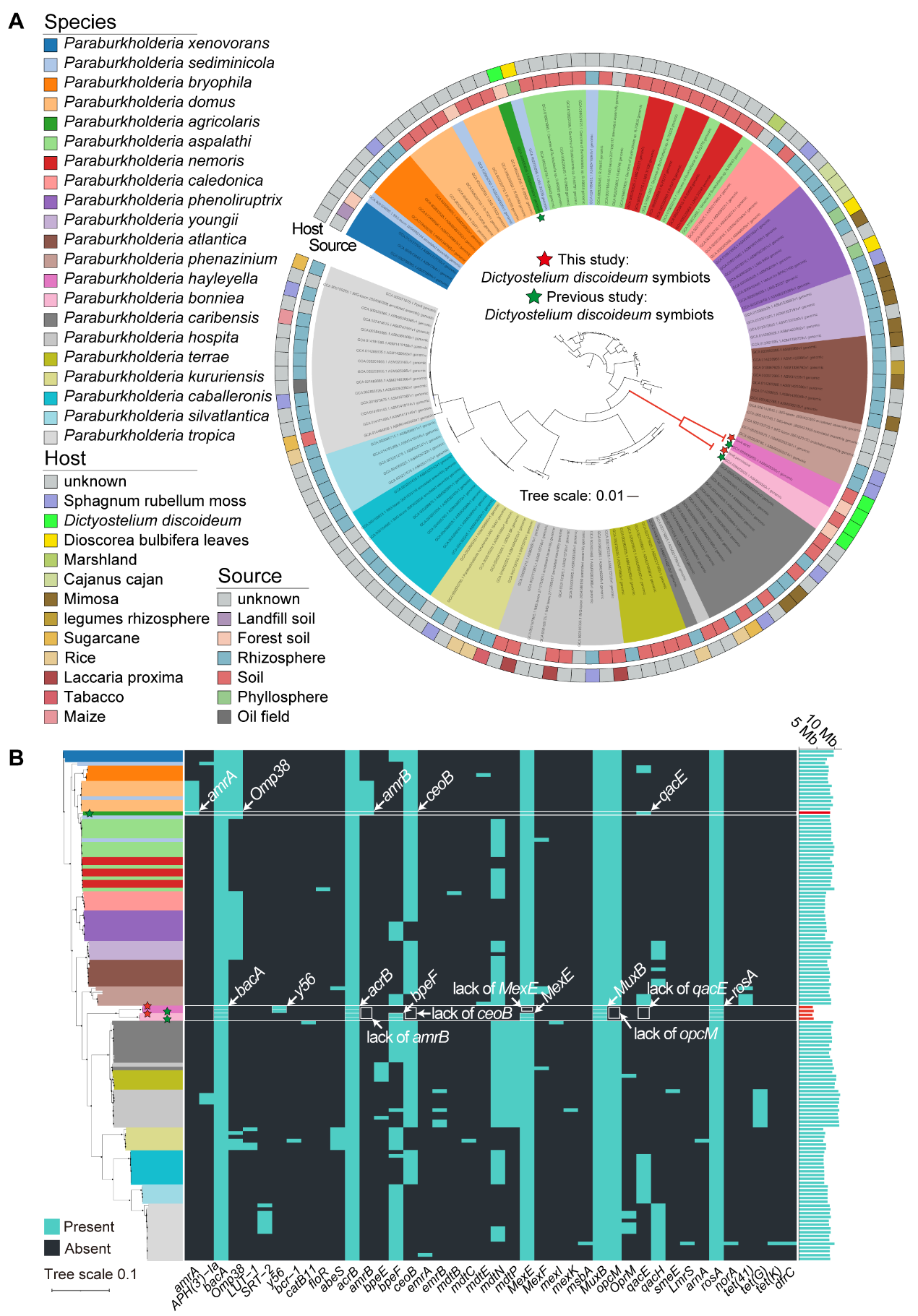
Figure 5. (A) Phylogenetic analyses of the two-assembled genomes bin4 and bin6. (B) Comparative genomics analyses of the resistance genes across 132 Paraburkholderia genomes. The blue bars represent the genome sizes of Paraburkholderia species.
我们发现土壤阿米巴携带具有高耐药性的临床人类病原体——嗜麦芽链球菌(S. maltophilia)。嗜麦芽链球菌被认为是医院环境中新出现的耐药病原体。为了评估阿米巴内源性嗜麦芽链球菌对人类健康的潜在风险,我们对bin7 MAG与NCBI数据库中的180个公开嗜麦芽链球菌基因组进行了比较分析。系统发育树分析表明,bin7属于Sm7谱系,并且与来自人类活动和人类呼吸来源的菌株非常接近。与自由生活的细菌相比,作为稳定共生菌的细菌在转变为细胞内生活方式时,其基因组会经历缩减。与P. hayleyella和P. bonniea相比,我们并未观察到阿米巴内源性S. maltophilia的基因组大小有显著减少。此外,我们在S. maltophilia中识别到较少的移动性相关基因,这表明S. maltophilia与D. discoideum阿米巴之间的相互作用仍在进行中,阿米巴宿主可能更多地作为促进S. maltophilia传播的临时介质。这些发现共同表明,与从其他环境中分离的菌株相比,阿米巴内源性S. maltophilia表现出显著的耐药性和毒力。基于高度相似的遗传特征,阿米巴内源性S. maltophilia可能具有在D. discoideum阿米巴和人类宿主内部生存的潜力。
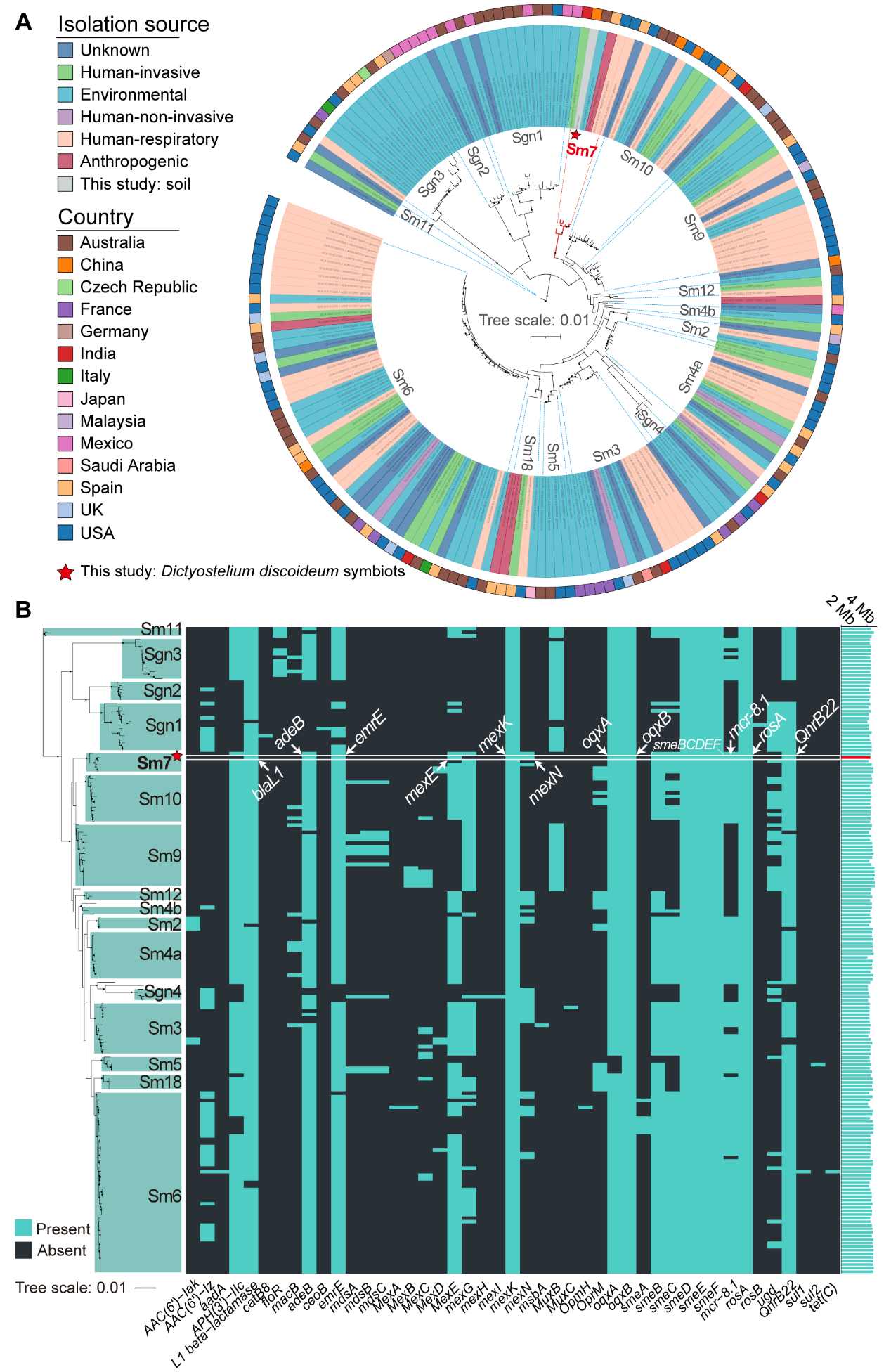
Figure 6. (A) Phylogenetic analyses of the assembled genome (bin7) of S. maltophilia. (B) Comparative genomics analyses of the resistance genes across 180 S. maltophilia genomes.
结论
与那些主要关注细菌的抗生素抗性研究不同,我们的研究揭示了土壤阿米巴——一个重要但通常被忽视的原生生物群体——实际上是环境抗生素和抗生素抗性基因的意外热点。这一发现显著地推进了我们对抗生素与微生物群落相互作用的理解,并提供了三个重要的新见解:首先,阿米巴显示出对高风险环境抗生素的高抗性,并能在细胞内积累大量抗生素;其次,原生生物可能在抗生素抗性基因的起源、转移和进化中扮演着关键角色;最后,从“同一健康”的角度来看,土壤原生生物在抗微生物抗性中的重要性不容忽视。因此,我们的研究建议将原生生物纳入抗生素抗性研究的范畴,并强调了它们在“同一健康”视角中的关键地位。
致谢
研究得到了国家自然科学基金(42030703和42277382)、广东省杰出青年自然科学基金(2023B1515020096)、南方海洋科学与工程广东省实验室(珠海)(SML2023SP218)和广东省自然科学基金(2023A1515011526)的支持。
论文链接:
Y Shi, M Liang, J Zeng, Z Wang, L Zhang, Z He, M Li, L Shu, G Ying. Soil Amoebae Are Unexpected Hotspots of Environmental Antibiotics and Antibiotic Resistance Genes, Environmental Science & Technology, 2024, https://url.scnu.edu.cn/record/view/index.html?key=9391b5dc5fc5c952dbcba6222b15a0f0.